空间组学技术可以同时测量细胞的空间位置和分子表达信息,是单细胞技术之后的最新一代测序技术。经过近几年发展,空间组学技术种类逐渐丰富,积累了大量复杂的生物数据。这些公开数据为生物学家提供了宝贵的资源,用以深入分析获得新的生物学知识;生物信息学家也可以基于公开数据来设计新颖计算方法。空间聚类(Spatial clustering)是目前解析组织微环境的主要手段,这类算法数量众多但缺乏全面定量分析。
近日,我院原致远联合中国科学院计算技术研究所赵屹研究员,建立了国际上首个空间组学聚类分析基准,研究成果以《空间转录组聚类基准研究》“Benchmarking spatial clustering methods with spatially resolved transcriptomics data”为题发表于《Nature Methods》期刊。
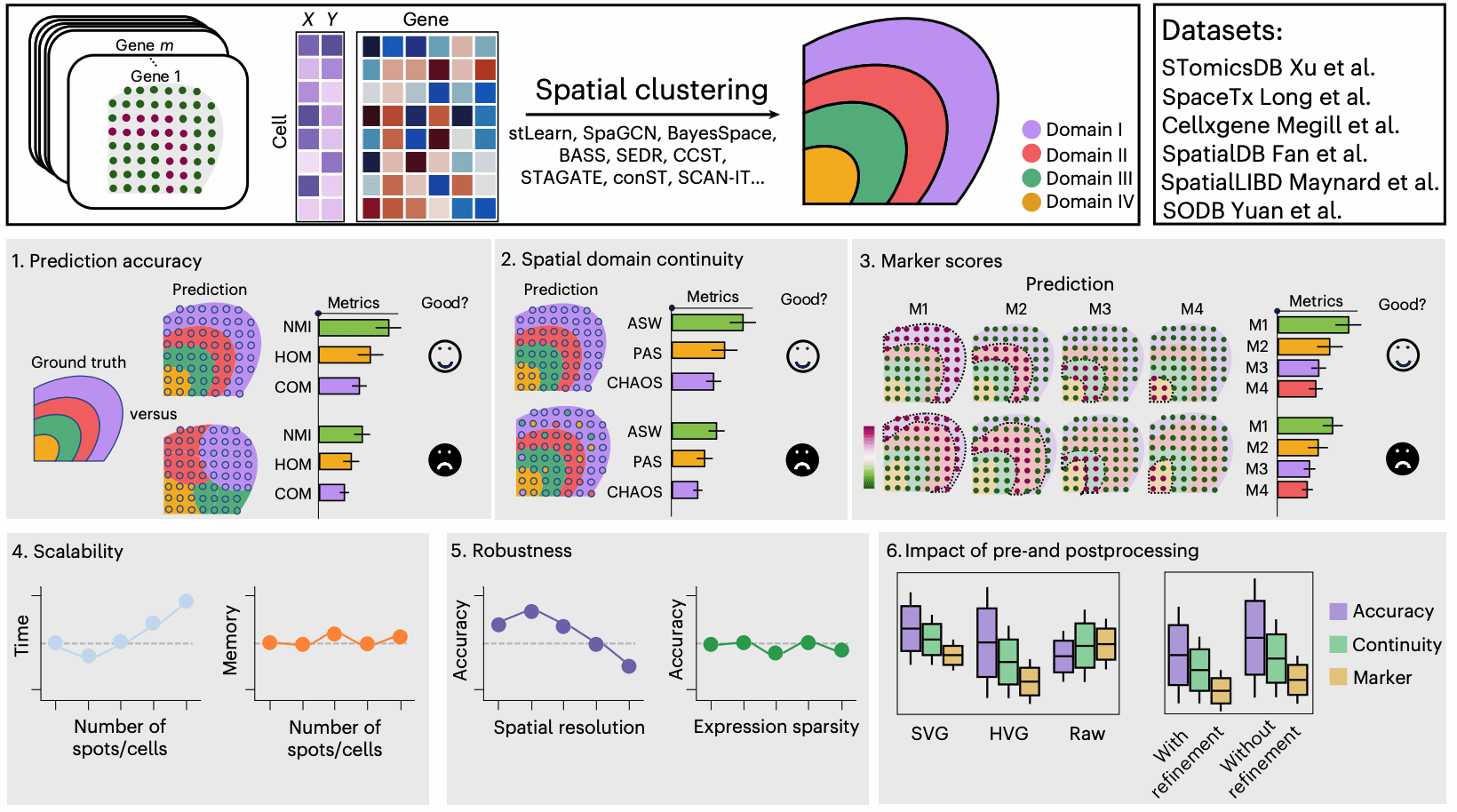
▲ 图1:SDMBench整体框架
该工作从理论层面解释了目前两大主流空间聚类算法(图卷积神经网络和概率图模型)的优劣势,从实践层面对目前13个主流算法进行了全面定量分析,包括准确性、生物标志物识别能力、拓展性、鲁棒性等,研究了预处理/后处理等因素对算法效果的影响,最终建立了国际上首个空间聚类算法基准SDMBench。研究发现没有一种通用方法适用于所有数据集,最优方法取决于数据本身特性(图2)。

▲ 图2:算法定量分析
该工作发现目前分析算法的一些局限性,包括难以识别较小和不连续空间域、难以分析大规模与多样本复杂实验设计的场景、对H&E等多模态数据应用不足等问题,并用了一个哺乳动物大脑衰老图谱数据作为例子,证明已有算法均会遇到时间/内存效率问题导致无法解决该问题。基于SDMBench的算法推荐结果,提出了分而治之的方法来结合已有算法解决该大规模任务(图3)。
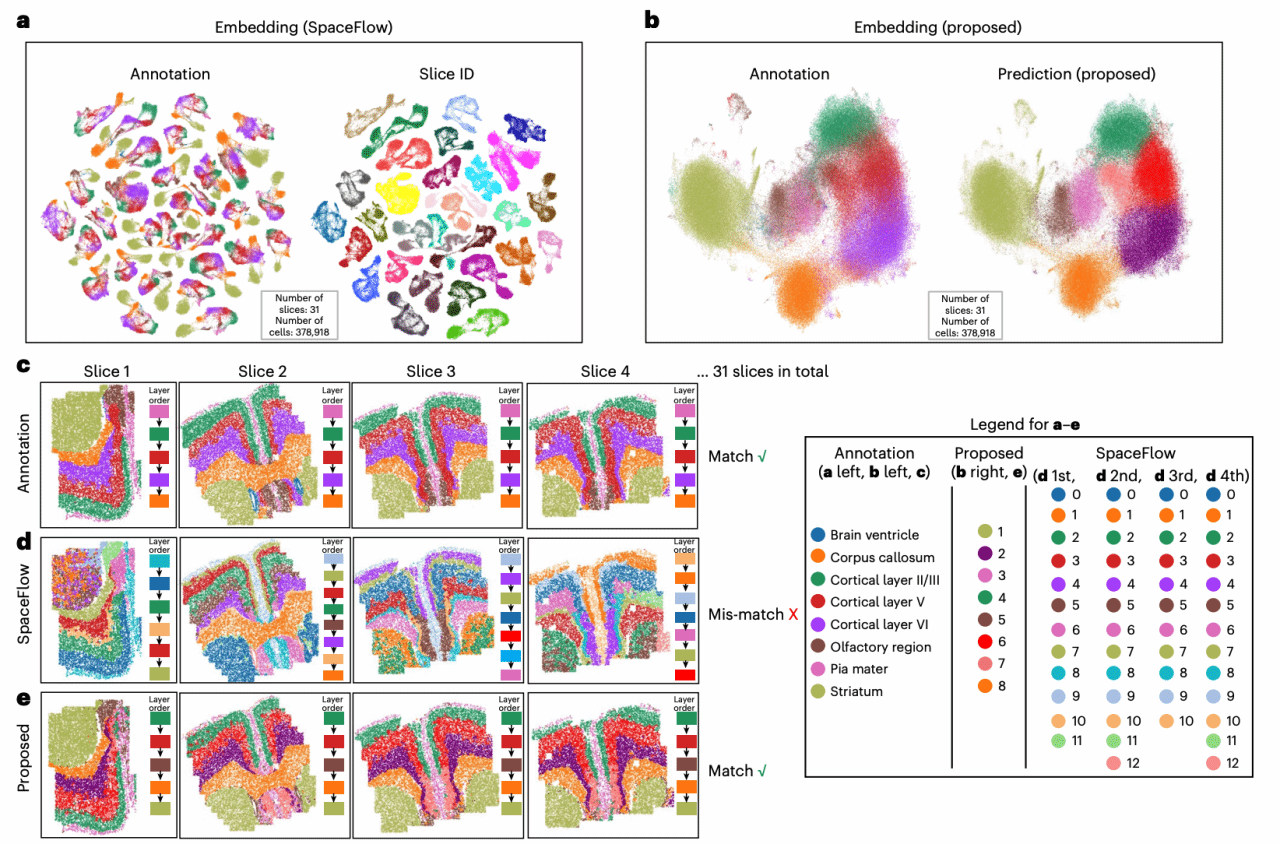
▲ 图3:分治算法解决大规模计算任务
综上,该工作建立了一个空间组学聚类问题的基准框架,为生物学家提供了一个全面的推荐机制以根据数据特征来选择最优算法,提供了一个具体案例展示了如何采用基准测试结果结合已有计算工具解决一个单一算法难以解决的大规模计算问题。该工作还提出了目前算法尚未解决的问题,从而启发了未来相关算法的研究方向。
实验室原致远(兼第一作者)和中国科学院计算技术研究所赵屹研究员为本文的共同通讯作者,中国科学院计算技术研究所博士生赵方圆为本文的共同第一作者,姚建华博士、赵宇博士、张孝勇博士、博士研究生林森林、硕士研究生崔岩为本课题做出重要贡献。
该课题得到国家自然科学基金、上海市晨光计划、上海市扬帆计划、腾讯AI Lab犀牛鸟计划、上海市重大专项等资助。
原文链接:https://www.nature.com/articles/s41592-024-02215-8