蛋白质是生命过程中的重要执行者。使用最广泛的蛋白质分析方法是面向大型蛋白质数据库搜索同源蛋白质,并从中推断查询蛋白质的特性,例如其功能。因此,同源蛋白质搜索被广泛应用于蛋白质功能预测、蛋白质结构预测、蛋白质-蛋白质相互作用预测等生物信息学方法。
远同源搜索挑战是识别结构相似但序列不相似的蛋白质对。与序列同源性不同,结构同源性可以在更长时间的进化过程中保留,因此提供了更多与功能相关的信息。而传统的蛋白质序列比对方法往往无法搜索出这些远同源对。
2024年3月30日,实验室朱山风研究员团队联合山东大学杨建益教授团队提出了基于蛋白质语言模型的最新远同源蛋白质搜索算法,研究成果以《PLMSearch:蛋白质语言模型为远程同源性提供了准确快速的序列搜索》“PLMSearch: Protein language model powers accurate and fast sequence search for remote homology”为题发表于《Nature Communications》期刊。
PLMSearch仅以序列作为输入,并使用蛋白质语言模型和Pfam序列分析来搜索同源蛋白质,这使得PLMSearch能够捕获隐藏在序列背后的远同源信息。PLMSearch主要包含三个步骤(图1 a-c)。(a)PfamClan。使用PfamClan过滤出共享相同Pfam Clan结构域的蛋白质对。(b)相似度预测。使用蛋白质语言模型为查询和目标蛋白质生成深度序列嵌入。随后,SS-predictor预测出所有查询-目标对之间的相似度。(c)得到搜索结果。最后,PLMSearch 筛选出PfamClan预过滤出的蛋白质对的相似度,根据相似度排序,并分别得到每个查询蛋白质的搜索结果。除此之外,对于搜索出的查询-目标对,我们使用PLMAlign生成局部或全局比对并计算比对分数(图1d)。
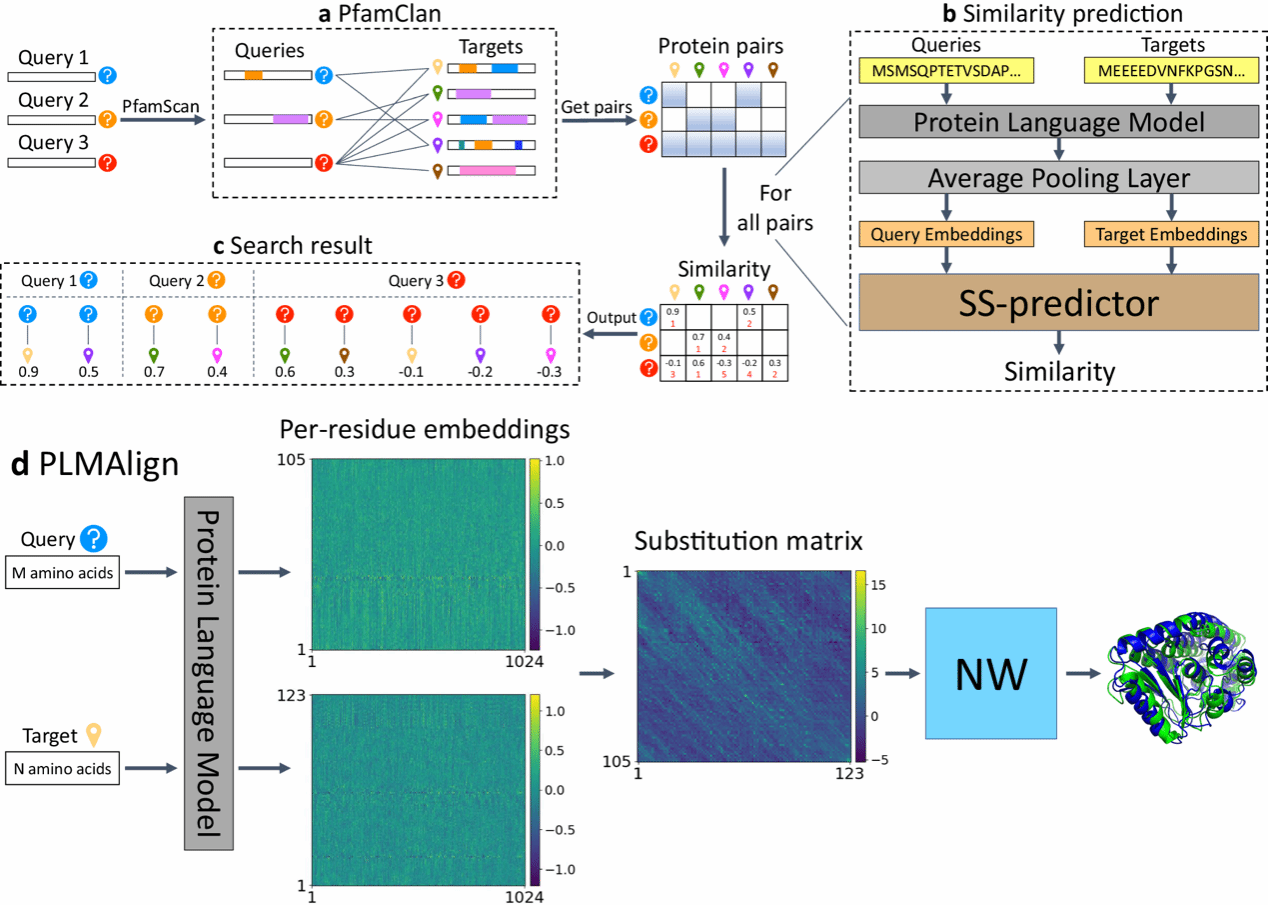
▲ 图1:PLMSearch + PLMAlign 整体框架
测试结果表明,PLMSearch始终是最好的搜索方法之一,并且兼顾了准确性和速度。具体来说,PLMSearch可以像MMseqs2一样在几秒钟内搜索数百万个查询-目标蛋白质对,但将灵敏度提高三倍以上,并且灵敏度接近最先进的结构搜索方法。
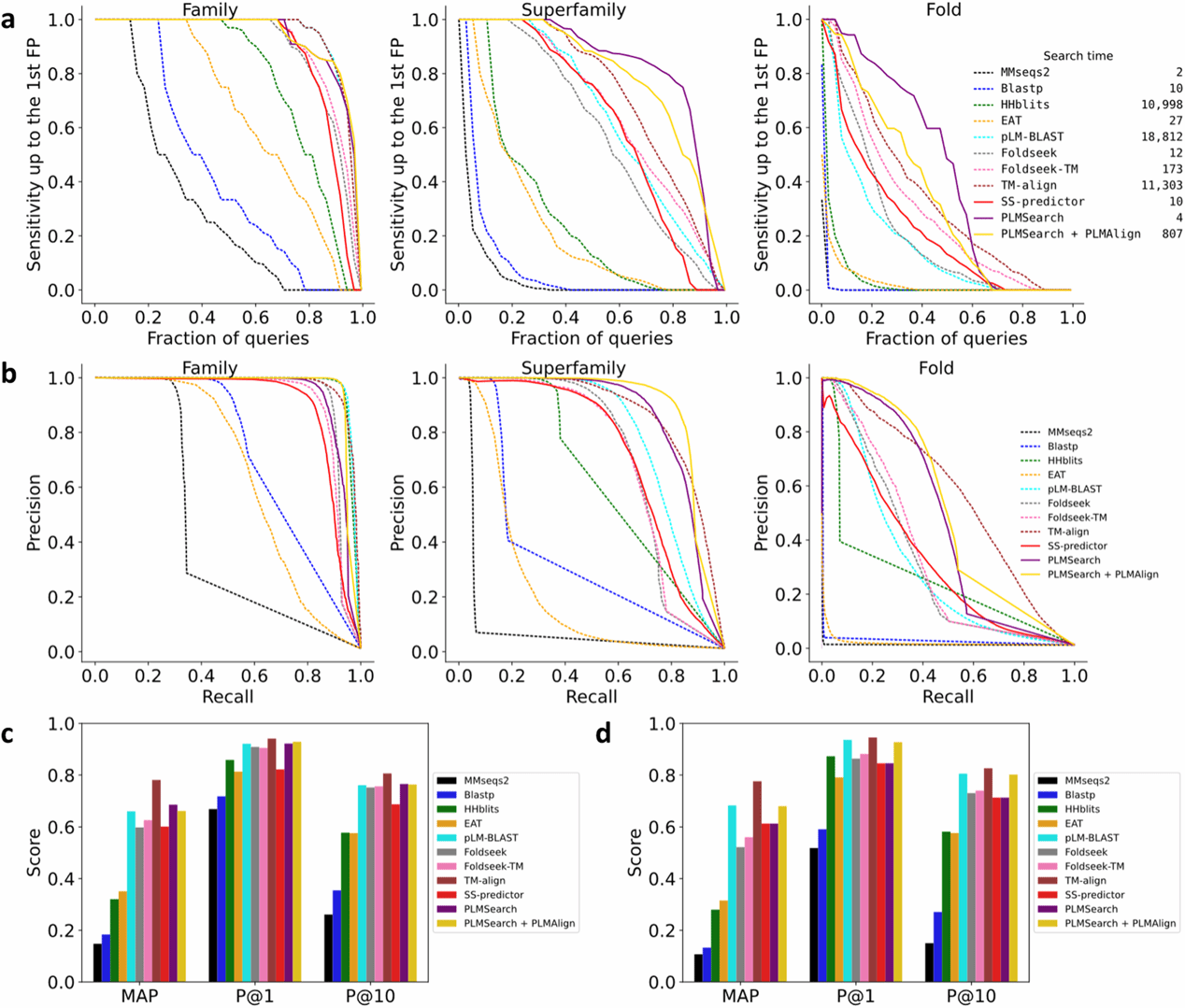
▲ 图2:PLMsearch 达到了与结构搜索方法相近的灵敏度
与简单的同源对相比,远同源对的同源性更难检测。在六种方法中,即使是最不敏感的方法MMseqs2和Blastp也能召回所有的简单对,但在召回远同源对时表现不佳。然而,在蛋白质语言模型的支持下,SS-predictor和PLMSearch能够召回大多数远同源对,并且召回率超过直接使用结构数据作为输入的Foldseek(图3)。
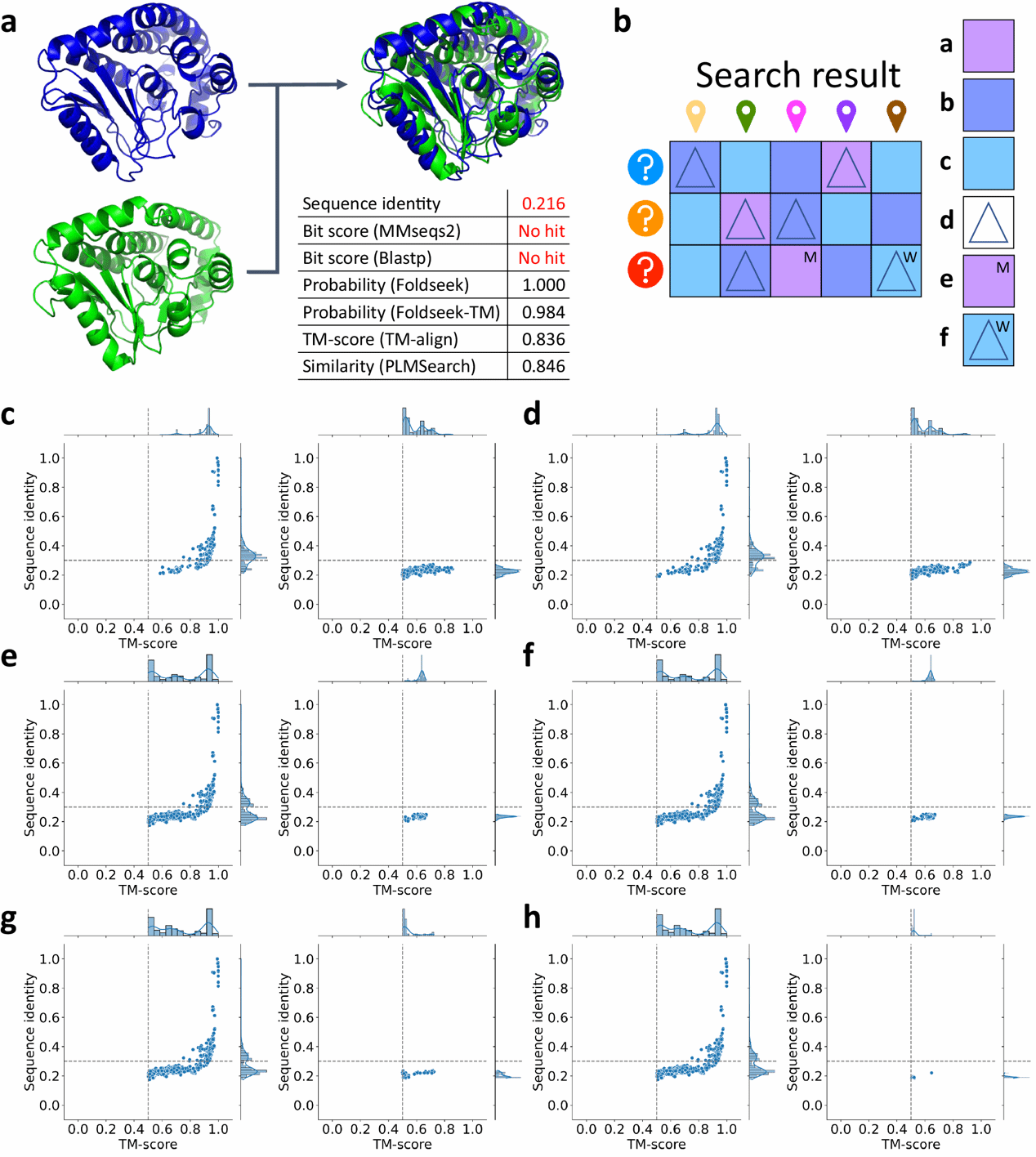
▲ 图3:PLMSearch 能够准确检测远同源蛋白质对
实验室硕士生刘炜是本研究的第一作者。实验室朱山风研究员和山东大学数学与交叉科学研究中心杨建益教授是本研究的通讯作者。上海交通大学熊毅副研究员,实验室王子叶博士后,游榕晖博士后(现为南开大学数据与统计科学学院特聘副研究员)等为本课题做出重要贡献。
本研究受到国家自然科学基金、上海市市级科技重大专项、北京智源人工智能研究院(BAAI)等的经费资助。
原文链接:https://www.nature.com/articles/s41467-024-46808-5