细胞命运由细胞内分子调控网络和细胞外微环境共同决定。单细胞组学技术可以测量细胞内的分子表达谱,但损失了空间位置信息。近年来,空间组学技术(Spatial Omics)的快速发展,使得实验人员可以在测量丰富的分子表达谱之外,额外获得空间位置信息,为解析组织微环境提供了条件。
主流空间组学技术包含空间转录组、空间蛋白质、空间代谢组等。随着技术的发展,大量的空间组学数据快速产生,并存储在各种异构数据平台中。在具体数据分析之前,研究者首先必须下载原始数据,然后将其正确处理为标准格式,这通常费时费力。
另一个问题是数据量,因为目前一些先进实验技术提供大视野和高空间分辨率(例如Stereo-seq、MERFISH等),处理这些数据需要大量内存和时间消耗。
复旦大学计算神经科学与类脑智能教育部重点实验室原致远青年副研究员与德州大学达拉斯分校张奇伟教授、腾讯人工智能实验室姚建华博士合作的最新研究成果给出了解决方案,支持用户快速浏览、可视化、读取、处理感兴趣的数据。
当地时间2月16日,相关研究成果以《SODB促进空间组学数据的全面探索》(“SODB facilitates comprehensive exploration of spatial omics data”)为题在《自然·方法》(Nature Methods)杂志上发表。
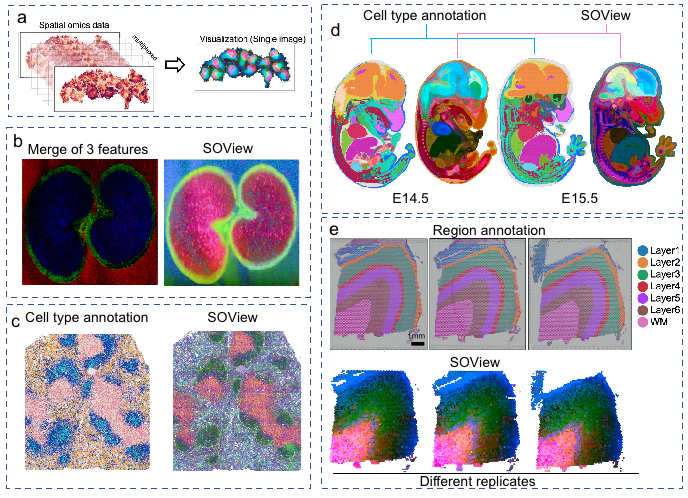
图2. SOView可视化模块
该数据库提供了来自26种空间组学技术,数据量超过5000万细胞(spot),所有数据均由标准流程处理为Anndata形式,可兼容SCANPY、Squidpy等多种分析软件。SODB还提供了多种数据分析和可视化模块,包括基因空间表达、细胞类型注释、基因表达比较、SOView可视化分析等(图2)。此外,还提供了配套Python工具包pysodb,仅用一行代码可以方便的读取数据,相比传统数据处理和读取方式,带来时间效率和内存效率的提升。以Slide-seq数据为例,时间效率提升160倍(传统方法读取需要19.04分钟,pysodb仅需7.16秒);内存效率提升549倍(传统方法需要21.97GB,pysodb仅需0.04GB峰值内存)。
目前,该数据库可通过以下网站公开访问:
[https://gene.ai.tencent.com/SpatialOmics/]或者Python包[https://github.com/TencentAILabHealthcare/pysodb]
本课题共同通讯作者是复旦大学计算神经科学与类脑智能教育部重点实验室原致远(兼第一作者)、腾讯AI Lab姚建华博士、德州大学达拉斯分校张奇伟教授;清华深研院潘文韬为本文共同第一作者;中科院计算所赵屹教授、清华深研院李秀教授、腾讯AI Lab赵轩、许志梦等合作者为本课题做出重大贡献。
本课题受到上海市“脑与类脑智能基础转化应用研究”市级重大科技专项 (No.2018SHZDZX01)、张江实验室、上海脑科学与类脑研究中心、高等学校学科创新引智计划(No.B18015)等经费资助。
原文链接:
https://www.nature.com/articles/s41592-023-01773-7

原致远,2022年6月于清华大学获博士学位,师从张奇伟教授。2022年9月加入复旦大学类脑智能科学与技术研究院,担任青年副研究员。主要从事生物信息学,特别是空间组学计算方法的研究与应用,主要涉及理论包括深度学习、统计建模及概率图模型,应用场景包括大规模脑时空图谱的构建、脑疾病及肿瘤微环境的时空建模。近年来在Nature Methods (2021、2023)、Nature Communications (2022a、2022b)、Nucleic Acids Research (2022)发表多篇论文。