细胞命运由细胞内分子调控网络和细胞外微环境共同决定。单细胞组学技术可以测量细胞内的分子表达谱,但损失了空间位置信息。近年来,空间组学技术(Spatial Omics)的快速发展,使得实验人员可以在测量丰富的分子表达谱之外,额外获得空间位置信息,为解析组织微环境提供了条件。
复旦大学计算神经科学与类脑智能教育部重点实验室原致远与德州大学达拉斯分校张奇伟教授、腾讯人工智能实验室姚建华博士合作的最新研究成果给出了空间组学数据中组织微环境的建模方案。
11月28日,相关研究成果以《SOTIP 是一种利用空间组学数据进行微环境建模的通用方法》(“SOTIP is a Versatile Method for Microenvironment Modelling with Spatial Omics Data”)为题在《自然·通讯》(Nature Communications)杂志上发表。

该研究通过最优传输理论构建了微环境之间的相互关系网络,将细胞分子表达谱的低维流形与空间局部拓扑特征联系起来,达到多个重要计算任务的同时分析,包括微环境异质性定量、空间域识别及差异微环境分析(图1)。SOTIP在多种空间转录组、蛋白组和代谢组数据的测试中展现出较好的准确性、稳定性和鲁棒性。
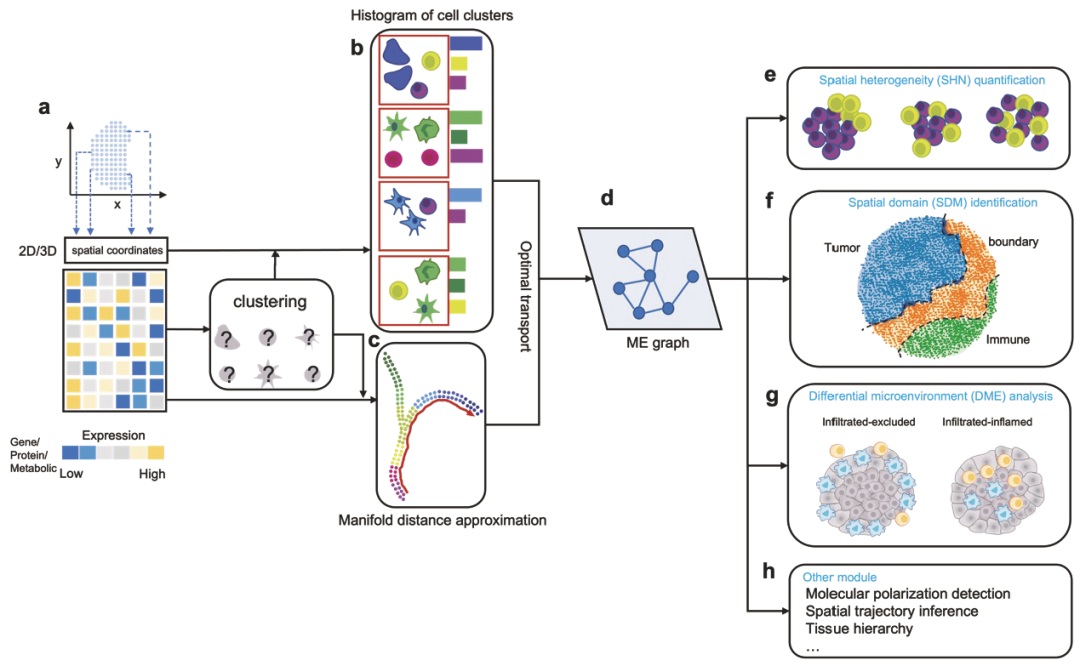
图1:SOTIP流程图
在空间异质性定量方面,研究人员在4i技术产生的Hela细胞系数据中精确地定量了细胞核膜和内质网膜附近的空间异质性,在10X Visium产生的斑马鱼肿瘤模型数据中识别了明确的肿瘤边界,在独立的小鼠大脑皮层数据中得到空间异质性在皮层深度轴上呈现一致的相关性。在空间域识别方面,SOTIP在多种空间蛋白组和转录组数据中呈现出较高的准确性,精确识别不同脑区及肿瘤结构,并能适用于三维空间数据。在差异微环境分析方面,SOTIP识别出三阴性乳腺癌亚型中特有的一种肿瘤-免疫微环境,并证实该微环境在病人队列中的富集性与病人预后显著相关。
综上,该工作开发了一种适用于多种空间组学数据的高扩展性多功能分析方法SOTIP。随着越来越多空间组学数据的产生,SOTIP会对解析其中分子和空间机理提供助力。
本课题共同通讯作者是复旦大学计算神经科学与类脑智能教育部重点实验室原致远(兼第一作者)、腾讯AI Lab姚建华博士、德州大学达拉斯分校张奇伟教授,清华大学李逸思博士为本文共同第一作者,清华大学师明磊博士、高军涛博士、腾讯AI Lab杨帆博士为本课题作出巨大贡献。本课题受到国家重点研发计划、国家自然科学基金等经费资助。
该成果是该课题组基于前期空间组学相关工作的又一拓展。前期工作提出了一个空间代谢组的计算分析方法,突破了现有方法的空间分辨率极限,实现单细胞级别的组织微环境分析。
该成果是该课题组基于前期空间组学相关工作的又一拓展。前期工作提出了一个空间代谢组的计算分析方法,突破了现有方法的空间分辨率极限,实现单细胞级别的组织微环境分析。
原致远,2022年6月于清华大学获博士学位。2022年9月加入复旦大学,担任青年副研究员。
主要从事生物信息学,特别是空间组学计算方法的研究与应用,主要涉及理论包括深度学习、统计建模及概率图模型,应用场景包括大规模脑时空图谱的构建、脑疾病及肿瘤微环境的时空建模。近年来在Nature Methods、Nature Communications、Nucleic Acids Research发表多篇论文。
原文链接:
https://www.nature.com/articles/s41467-022-34867-5