共病指的是一名患者身上非随机同时出现的两种或两种以上的疾病。与单一疾病相比,共病通常会降低患者的生活质量、增加患者的死亡率和经济支出。共病通常是环境和遗传的共同作用结果,其机制非常复杂。在过去十几年中,全基因组关联研究(GWAS)发现了少数共病之间存在共享的遗传风险位点和相似的遗传结构。
但是,由于缺乏大规模疾病的诊断和遗传数据,更加普适的共病遗传模式尚不清楚。过去十几年,英国生物样本库 (UK Biobank) 收集了大约 50 万人的电子病历和基因组数据,使得整合诊断数据与基因组数据以探索常见疾病的共病谱及其遗传模式成为可能。
近日,复旦大学计算神经科学与类脑智能教育部重点实验室赵兴明教授和陈靖祺青年副研究员团队,分析了常见疾病的共病谱及其多层次的遗传风险,发现共病网络中的hub疾病和分子或许是治疗共病的关键因素。
2021年7月5日,这一研究成果以“A global overview of genetically interpretable multimorbidities among common diseases in the UK Biobank”为题,在线发表于Genome Medicine期刊。
该研究基于英国生物样本库(UK Biobank)的电子病历数据,发现了438种常见疾病之间的11,285对共病。除影响相同生理系统的共病外,该研究还发现了41组跨生理系统的共病趋势,其中代谢疾病跨生理系统的共病率最高。并且大部分跨生理系统的共病趋势都是由少数的几种疾病介导的,例如:营养和心理、关节、脊椎系统之间的共病主要是由肥胖(Obesity)介导的。
研究人员结合英国生物样本库(UK Biobank)的全基因组关联分析的汇总统计数据,通过post-GWAS分析,在位点、网络和总体遗传结构水平上,发现了接近一半(47%)的共病可以由遗传因素解释,并且这些遗传可解释的共病在生理系统上表现出不同的遗传模式,即影响相同生理系统的共病倾向于由位点水平的遗传因素解释,而影响不同生理系统的共病倾向于由网络水平的遗传因素解释。这一结果表明了影响相同和不同生理系统的共病可能存在不同的生物学起源─前者倾向于直接来源于多效性位点,后者倾向于间接来源于相同或相似的生物功能。
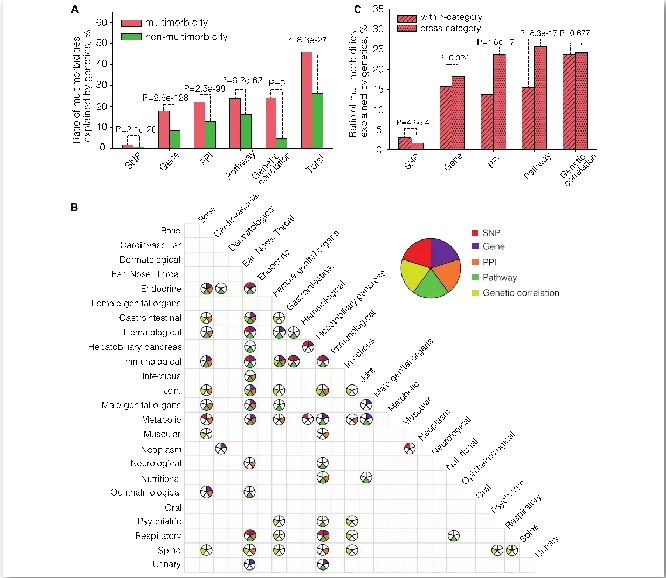
图1. 遗传可解释的共病:
A.通过单核苷酸多态性(SNP)、基因(gene)、蛋白相互作用(PPI)、生物学通路(pathway)和遗传相关性(genetic correlation)解释的共病和非共病比例。
B.不同遗传因素显著解释的共病类别。每个圆被分成了5份,分别表示五种遗传因素。每个圆被颜色填充的部分表示该种遗传因素可以显著地解释该类别的共病。没有圆形的地方表示没有一种遗传因素可以显著地解释该类别的共病。
C.生理系统内和生理系统间可以通过单核苷酸多态性(SNP)、基因(gene)、蛋白相互作用(PPI)、生物学通路(pathway)和遗传相关性(genetic correlation)解释的共病比例。
研究人员通过对共病共享的分子和网络进行功能分析,发现了少数的遗传元件可以解释很大一部分的共病,而这些常见的遗传元件主要与细胞免疫、蛋白质代谢和基因沉默等功能有关。研究人员将“遗传位点解释的共病”和“分子网络解释的共病” 进一步构建为两个遗传可解释的共病网络,并通过网络结构分析,发现了这两个共病网络中均存在明显的模块结构,并且找到了模块内部的hub疾病和分子。这些hub疾病和分子可以帮助我们理解共病的病因。
例如:烟草依赖与5种呼吸系统疾病(肺气肿,脓胸、呼吸衰竭、慢性阻塞性肺疾病、肺炎)、3种心血管疾病(动脉粥样硬化、主动脉瘤与夹层、周围血管疾病)以及肺癌形成一个共病模块,其中烟草依赖是这个模块的hub疾病。该模块中最常见的基因是IREB2和CHRNA3,其中IREB2编码一种调节铁代谢的铁反应元件结合蛋白(IRP),CHRNA3编码神经元烟碱乙酰胆碱受体。该结果表明,铁代谢和神经元烟碱乙酰胆碱受体或许是研究烟草依赖与这些疾病共病的首选靶点。
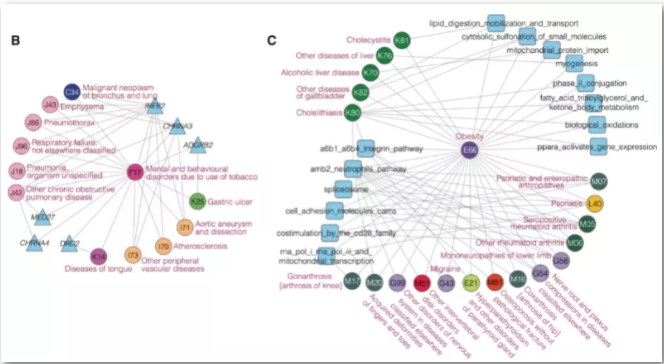
图2. 遗传可解释的共病网络研究案例:圆形、三角形和正方形分别代表疾病、基因和生物学通路。圆形(疾病)的颜色对应于疾病的类别。
B. LG-module5中hub疾病-F17烟草依赖的共病,以及它们共享的基因。
C. NG-module1和NG-module6中hub疾病-E66肥胖的共病及其共享的生物学通路。图中只显示了解释共病对数最多的5条通路。
该研究首次整合相同人群的电子病历和基因组数据,对常见疾病之间的共病关系及其遗传风险进行了系统分析。该研究阐明了生理系统内和生理系统间的共病趋势及共病遗传模式,并指出hub疾病和相同或相似的生物分子可能是治疗共病的关键。
研究人员已经建立了一个在线数据库,方便其他研究人员和医生浏览、搜索或下载这些共病:
https://multimorbidity.comp-sysbio.org
复旦大学计算神经科学与类脑智能教育部重点实验室博士生董桂英是该论文的第一作者,赵兴明教授和陈靖祺青年副研究员为共同通讯作者。该研究得到上海市“脑与类脑智能基础转化应用研究”市级科技重大专项资助、国家重点科研项目、国家自然科学基金、高等学校学科创新引智计划、上海市自然科学基金、张江实验室、上海脑科学与类脑研究中心的经费支持。
原文链接:
https://genomemedicine.biomedcentral.com/articles/10.1186/s13073-021-00927-6