重叠群分箱(contig binning)是宏基因组数据分析中的关键步骤,其目标是将来自相同基因组或密切相关基因组的重叠群聚集到同一个基因组分箱(bin)中。通过宏基因组组装和分箱得到的基因组箱,通常被称为宏基因组组装基因组(metagenome-assembled genomes,简称MAGs),其质量直接影响着新微生物基因组的发现以及微生物群落结构和功能的分析等后续研究的可靠性。因此,开发高效且高性能的重叠群分箱算法成为当前宏基因组研究中的一个重要的问题。
2024年1月17日,实验室朱山风研究员团队在《自然·通讯》Nature Communications期刊上发表了题为《基于多视图对比学习实现宏基因组重叠群的高效分箱》(Effective binning of metagenomic contigs using contrastive multi-view representation learning)的研究论文。

朱山风研究员团队提出了一种基于多视图对比学习的分箱方法,称为COMEBin。COMEBin充分利用数据增强,生成每个重叠群的多个片段(视图),并通过对比学习获得异构特征(例如序列覆盖率和k-mer分布)的高质量嵌入。实验结果表明,在多个模拟和真实数据集上,COMEBin相较于当前最先进的分箱方法表现更优,尤其是在从真实环境样本中恢复近乎完整的基因组方面。
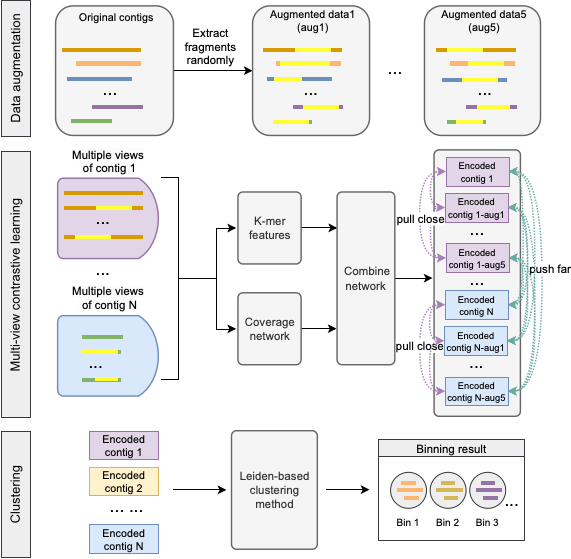
▲图1:COMEBin框架
当集成到宏基因组分析流程中时,COMEBin 的表现优于其他分箱方法,包括恢复潜在致病性抗生素耐药细菌 (PARB) 和含有潜在生物合成基因簇 (BGC) 的中等或更高质量的分箱。
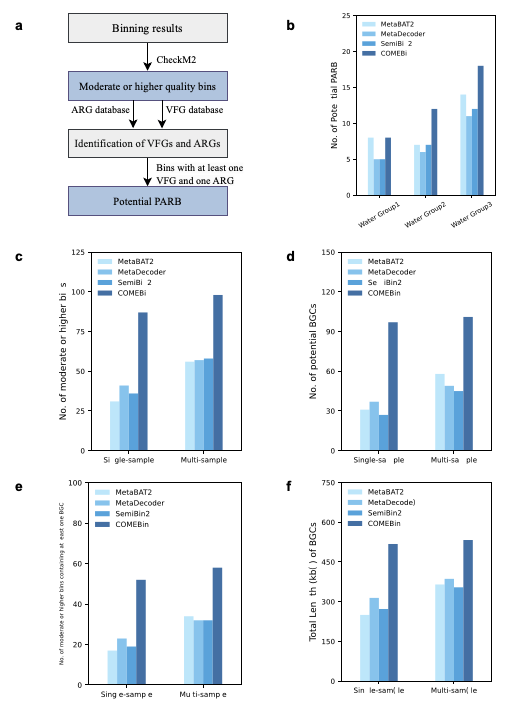
▲图2:恢复潜在致病性抗生素耐药细菌(PARB)以及
含有潜在生物合成基因簇(BGC)的中等或更高质量的基因组箱
复旦大学计算神经科学与类脑智能教育部重点实验室博士后王子叶是本研究的第一作者,朱山风研究员是本研究的通讯作者。美国南加州大学孙丰珠教授是本研究的合作者。本研究受到国家自然科学基金、上海市市级科技重大专项、中国博士后科学基金等的经费资助。
原文链接:https://www.nature.com/articles/s41467-023-44290-z