空间组学技术在保留细胞空间位置的同时,可以精确测量分子表达信息,为理解生理或病理状态下的组织结构及微环境提供了前所未有的机会。空间聚类是识别组织微环境的主要计算手段,也是目前空间组学计算领域中热点问题。目前空间聚类算法包括两大类:基于图神经网络的算法和基于概率图模型的算法。这两大类算法均依赖空间细胞网络的构建,在大规模数据下,图神经网络存在显存溢出问题,概率图模型存在优化效率低的问题,导致目前算法无法轻易拓展到超百万级别数据分析。随着多时间点、多样本、多条件等复杂实验设计的数据集越来越多地发布,样本间整合/比较分析难题也逐步显露并亟待解决。

为此,实验室原致远建立了一种新的空间组学微环境表征和聚类算法MENDER,解决了空间组学以上两大难题。1月5日,相关成果以《MENDER:一个快速且可扩展的空间组学组织结构识别方法》(“MENDER: fast and scalable tissue structure identification in spatial omics data”)为题,发表于《自然·通讯》Nature Communications期刊。
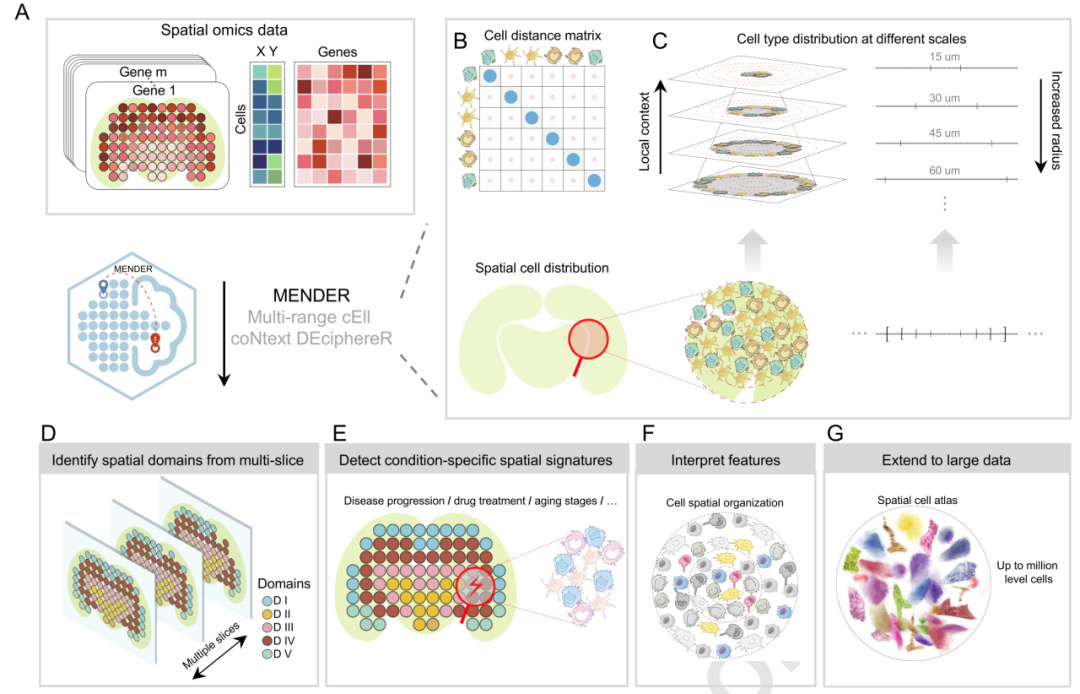
▲图1:MENDER整体框架
区别于过去算法,MENDER算法基于多种空间组学平台之间细胞邻域结构的保守性观测,提出一种新型非参数化卷积核,在细胞邻域信息提取步骤上绕过了高时间和显存占用的参数更新步骤,从而大幅度降低内存/显存消耗,提高时间效率。在3种不同有监督任务上,性能超越目前主流图网络学习算法。在无监督场景下,MENDER在3种不同组织的跨平台空间组学数据中,MENDER的性能指标均超越或与目前最优算法持平,而时间消耗大幅减少,在25分钟左右即可完成73万细胞大脑空间图谱的分析(无需GPU,Xeon(R) Gold 5218R CPU @ 2.10 GHz 80 cores)。MENDER可以在跨样本间得到天然对齐的微环境标签,无需额外对齐操作。
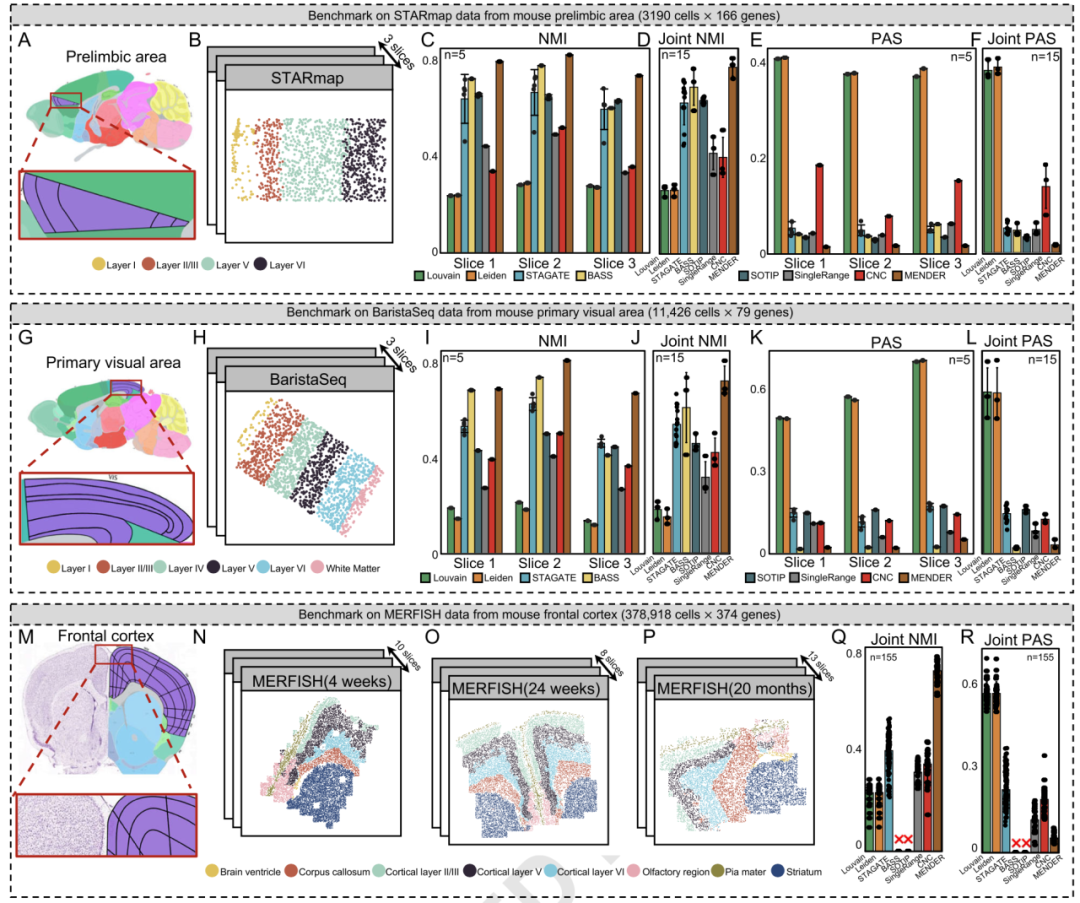
▲图2:MENDER算法性能
用MENDER分析大脑衰老空间细胞图谱,成功识别出两类原文未标注出的大脑结构,其中一个大脑结构的细胞空间组成与衰老过程密切相关,只在幼年(4周龄)小鼠中出现。这一发现可以在MENDER跨切片整合和比较模块中直观可视。MENDER提供的微环境解释性分析也揭示了造成差异的因素主要是少突胶质细胞亚型的区域性富集。

▲图3:MENDER识别衰老相关结构
最后,对一个大型三阴性乳腺癌病人队列数据,采用MENDER识别出的空间结构对病人进行表征,能成功区分开三种不同预后的病人分群,而这三种分群用传统单细胞为单元的分析无法区分。这一例子说明了多细胞组成的空间结构或微环境对疾病发展的预测作用,同时加强了大规模数据的整合分析在空间组学大数据时代的生物学和医学价值。
复旦大学计算神经科学与类脑智能教育部重点实验室原致远为本文的唯一作者。该课题得到国家自然科学基金、上海市晨光计划、上海市启明星项目(扬帆专项)、腾讯AI Lab犀牛鸟计划、上海市市级科技重大专项等资助。
原文链接:https://www.nature.com/articles/s41467-023-44367-9